Zusammenfassung
In diesen Zeiten Molekularbiologie zu betreiben ist aufregend. Es bedeutet »Gentechnik« und »Klonieren« und hat etwas Göttliches. Beim einen Teil der Bevölkerung wird man, wenn man verrät, womit man seinen lieben langen Arbeitstag verbringt, grenzenlose Bewunderung hervorrufen, beim anderen grenzenlose Ablehnung – man sollte sich daher genauestens überlegen, mit wem man es gerade zu tun hat, bevor man den Mund aufmacht. Am besten, man erwähnt keiner der Gruppen gegenüber, mit wieviel Problemen und Frust man in Wahrheit täglich kämpft, weil der erste Teil dann desillusioniert wäre und der zweite, vielleicht zurecht, unweigerlich die Frage stellen würde: »Wozu machst du das dann überhaupt?«
You have full access to this open access chapter, Download chapter PDF
Gib nach dem löblichen Verlangen, von vorn die Schöpfung anzufangen! Zu raschem Wirken sei bereit! Da regst du dich nach ewigen Normen durch tausend, abertausend Formen, und bis zum Menschen hast du Zeit. (Goethe, Faust)Footnote 1
In diesen Zeiten Molekularbiologie zu betreiben ist aufregend. Es bedeutet „Gentechnik“ und „Klonieren“ und hat etwas Göttliches. Beim einen Teil der Bevölkerung wird man, wenn man verrät, womit man seinen lieben langen Arbeitstag verbringt, grenzenlose Bewunderung hervorrufen, beim anderen grenzenlose Ablehnung – man sollte sich daher genauestens überlegen, mit wem man es gerade zu tun hat, bevor man den Mund aufmacht. Am besten, man erwähnt keiner der Gruppen gegenüber, mit wieviel Problemen und Frust man in Wahrheit täglich kämpft, weil der erste Teil dann desillusioniert wäre und der zweite, vielleicht zurecht, unweigerlich die Frage stellen würde: „Wozu machst du das dann überhaupt?“
Am meisten, muss ich gestehen, gefallen mir in diesem Zusammenhang all die nutzlosen Diskussionen darüber, ob man Menschen klonieren solle oder nicht. Eigentlich ist's ja mehr eine Art Pingpong: Einer macht einen blödsinnigen Vorschlag und die halbe (Medien-) Nation erklärt, weshalb das die Klonierung von Menschen keinesfalls rechtfertige. Viel Lärm um nichts, wie mir scheint, ich habe jedenfalls noch immer nicht verstanden, zu welchem Vorteil es mir gereichen könnte, mich klonen zu lassen. Wozu zwanzig- oder fünfzigtausend Euro zahlen für einen kleinen Schreihals, dessen einzige Gemeinsamkeit mit mir darin besteht, dass er so aussieht wie ich vor dreißig Jahren, wenn ich auf klassischem Wege zu einem ähnlichen Ergebnis kommen kann, zum Preis von einem Blumenstrauß für meine Frau und einem fernsehfreien Abend. Mehr Spaß macht's übrigens auch.
Das Beispiel zeigt recht gut, vor welchem Problem die Molekularbiologie derzeit steht. Angeregt von mehr oder weniger spektakulären Berichten im Fernsehen hat jeder seine eigenen, zumeist weit übertriebenen Heils- oder Unheilsvorstellungen zu diesem Thema. Die Realität nimmt sich dagegen ziemlich niederschmetternd aus. Der Molekularbiologe, auch Molli genannt, hantiert die meiste Zeit mit winzigen Mengen zumeist klarer, farbloser Lösungen – keine Spur vom wildgewordenen Forscher, wie man ihn aus den Filmen kennt, der inmitten von wabernden, dampfenden, knallbunten Flüssigkeiten steht und dabei offensichtlich viel Spaß hat. Der Molli feilt an Molekülen herum, von deren Existenz ihn viele Lehrbücher zu überzeugen versuchen, obwohl er in der Praxis von ihnen kaum je mehr als einen fluoreszierenden Fleck im Agarosegel zu sehen bekommt. Irgendwie scheint jeder Arbeitsgang drei Tage zu dauern, und weit und breit winkt kein Nobelpreis.
Molekularbiologie ist vor allem Voodoo – mal klappt alles, meist klappt nix. Über den Ausgang eines Experiments scheinen recht seltsame Parameter zu entscheiden, die eigentlich selbst zum Gegenstand der Forschung gemacht werden sollten – das letzte große Tabu der Wissenschaft. Ich persönlich bin aufgrund von empirischen Daten zu der Überzeugung gekommen, dass man den Ausgang eines Experiments aus dem Quotienten aus Luftdruck und der verbleibenden Anzahl von Schmierblättern in der Schublade, potenziert mit dem Biorhythmus-Index von Omas Hund ermitteln kann. Bis es mir gelungen ist, dies experimentell zu belegen, werde ich mich allerdings auf die klassischen Erklärungsschemata beschränken müssen, mit denen dieses Gewerbe zugegebenermaßen schon recht weit gekommen ist.
Fangen wir an.
1.1 Das Substrat der Molekularbiologie, oder: Molli-World für Anfänger
War es ein Gott, der diese Zeichen schrieb, die mir das innre Toben stillen, das arme Herz mit Freude füllen und mit geheimnisvollem Trieb die Kräfte der Natur rings um mich her enthüllen?
Molekularbiologie, das ist keineswegs die Biologie der Moleküle, wie man annehmen würde – für diese Abteilung ist die Biochemie zuständig –, sondern die Welt der Leute, für die das Leben eine große DNA ist. Es ist eine kleine, eingeschworene Welt, die sich gerne nach allen Seiten hin abgrenzt, von den Zoologen, von den Botanikern, von den Proteinbiochemikern. Das kommt vermutlich daher, dass die wenigsten von ihnen Biologen sind – und diejenigen, die es sind, würden es am liebsten leugnen. Das war schon immer so und hat sich bis heute nur wenig geändert. Wenn Sie ebenfalls keiner sind, wissen Sie nun, dass Sie sich in bester Gesellschaft befinden. Das bisschen, das man für diese Arbeit wissen muss, lässt sich leicht nachholen, Sie werden sehen.
Die Molekularbiologie beschäftigt sich mit den Nucleinsäuren, von denen es zwei Arten gibt: DNA und RNA . Ersteres Kürzel steht für Desoxyribonucleinsäure, auf Englisch desoxyribonucleic acid (daher die Abkürzung), Letzteres für Ribonucleinsäure. Der chemische Unterschied zwischen den beiden ist gering, sie sind beide Polymere, die aus jeweils vier Bausteinen zusammengesetzt sind, Desoxynucleotide im einen Fall, Nucleotide im anderen.
Nucleotide bestehen aus einem Basenanteil (Adenin, Cytosin, Guanin oder Uracil), einem Zuckeranteil (Ribose) und einem Phosphatrest, wobei zwei Nucleotide jeweils durch eine Phosphat-Zucker-Bindung miteinander verknüpft sind. Auf diese Weise kann man ein Nucleotid ans nächste hängen und bekommt so eine lange Kette, ein Polynucleotid, das man als RNA bezeichnet. DNA ist fast genauso aufgebaut, nur eben aus Desoxynucleotiden, das heißt: statt Ribose wird 2'-Desoxyribose verwendet, außerdem findet man statt Uracil Thymin (Abb. 1.1).
Wer sich für die Details interessiert, sollte an diesem Punkt zu einem Lehrbuch der Biochemie, der Zellbiologie oder der Genetik greifen. Ich will mich stattdessen auf einige wenige Aspekte beschränken, die für die Laborpraxis von Bedeutung sind:
-
„Desoxynucleotid“ ist ein Zungenbrecher, den jeder offensichtlich umgeht, wo er nur kann, im Laborgebrauch spricht man daher einfach von Nucleotiden, obwohl man zumeist die Desoxy-Variante meint.
-
die Namen der Nucleotide sind den Namen der Basen, durch die sie sich unterscheiden, entlehnt, sie heißen Adenosin , Cytidin , Guanosin , Thymidin und Uridin . Viele werfen Base und Nucleotid durcheinander, für die Praxis macht das kaum einen Unterschied. Die Abkürzungen lauten A, C, G, T und U. Mit diesen fünf Buchstaben werden alle DNA- und RNA-Sequenzen dieser Welt verewigt.
-
die Phosphatgruppe des Nucleotids hängt am 5'-C-Atom des Zuckeranteils. Die Nucleinsäuresynthese beginnt mit einem Nucleotid, an dessen 3'-OH-Gruppe (das ist die Hydroxylgruppe am 3'-C-Atom) eine Phosphoesterbindung mit der Phosphatgruppe des nächsten Nucleotids geknüpft wird. An die 3'-OH-Gruppe des eben angehängten Nucleotids kann ein weiteres Nucleotid angehängt werden und so weiter und so fort; man sagt daher, dass die Synthese in 5' → 3'-Richtung abläuft, weil zu jedem Zeitpunkt ein 5'-Ende und ein 3'-Ende existiert und neue Nucleotide immer ans 3'-Ende gehängt werden. Alle Sequenzen dieser Welt sind immer in der 5' → 3'-Orientierung angegeben und man sollte darauf verzichten, diese Konvention durchbrechen zu wollen, wenn man nicht ein heilloses Chaos stiften will.
-
Existiert keine 3'-OH-Gruppe, kann kein neues Nucleotid angehängt werden. In der Natur kommt dieser Fall praktisch nicht vor, im Labor dagegen ist er von großer Bedeutung für die Sequenzierung. Zusätzlich zu den vier (2'-Desoxy-)Nucleotiden werden in diesem Fall bei der DNA-Synthese auch 2',3'-Didesoxynucleotide eingesetzt. Sie können ganz normal an das 3'-Ende eines Polynucleotids angehängt werden, weil ihnen aber die 3'-OH-Gruppe fehlt, kann die DNA nicht weiter verlängert werden. Dieses Prinzip ist für das Verständnis der Sequenzierung von eminenter Bedeutung, darauf wird in einem späteren Kapitel nochmals eingegangen.
-
Jedes Nucleotid oder Polynucleotid, das am 5'-Ende eine Phosphatgruppe trägt, kann mit Hilfe einer DNA-Ligase an das 3'-Ende eines anderen Polynucleotids gebunden (ligiert) werden. Auf diese Weise lassen sich auch größere DNAs miteinander verknüpfen. Ohne die Phosphatgruppe läuft dagegen gar nichts, durch Entfernen des Phosphatrests mit einer Phosphatase kann man daher Ligationen gezielt verhindern.
-
Zwei Nucleotide, die miteinander verknüpft sind, bezeichnet man als Dinucleotid, drei als Trinucleotid, sind es mehr, spricht man von Oligonucleotid , sind's sehr viele, bezeichnet man das Ganze als Polynucleotid. Die Grenze zwischen Oligo- und Poly- ist nicht genau definiert, in der Praxis wird man Nucleinsäuren von weniger als 100 Nucleotiden Länge als Oligonucleotid bezeichnen.
-
Egal ob Mono-, Di-, Oligo- oder Poly-, es handelt sich immer um ein einziges Molekül, weil bei jeder Verlängerung eine kovalente Bindung zwischen den beiden beteiligten Molekülen geknüpft wird. Die Länge des dabei entstehenden Lindwurms spielt keine Rolle, auch eine Nucleinsäure von 3 Millionen Nucleotiden Länge ist ein einziges Molekül.
-
Polynucleotide haben eine wunderbare und bedeutende Eigenschaft: Ihre Basenanteile paaren sich gerne mit anderen Basenanteilen. Dabei paart sich immer Cytosin mit Guanin und Adenin mit Thymin (bei DNA) bzw. Uracil (bei RNA), andere Kombinationen funktionieren nicht. Je mehr Basen miteinander paaren, desto stabiler wird diese Verbindung, weil bei jeder Paarung Wasserstoffbrückenbindungen zwischen den Basen gebildet werden, deren Kräfte sich addieren. Eine Sequenz, die perfekt mit einer anderen Sequenz paart, bezeichnet man als komplementär. Komplementär heißt dabei nicht etwa identisch, sondern lediglich zusammenpassend, mit anderen Worten: Die Nucleinsäuresequenzen zweier komplementärer Nucleinsäuren sind völlig verschieden! Ein Beispiel soll das verdeutlichen. Betrachten wir dazu den unteren Strang aus einem beliebigen Stückchen doppelsträngiger DNA:
 Genau genommen ist diese Schreibweise falsch, weil die beiden DNA-Stränge gegenläufig zueinander angeordnet sind. Korrekterweise
müsste man die obige Sequenz folglich so darstellen
Genau genommen ist diese Schreibweise falsch, weil die beiden DNA-Stränge gegenläufig zueinander angeordnet sind. Korrekterweise
müsste man die obige Sequenz folglich so darstellen
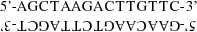 doch sind unsere Textverarbeitungsprogramme hierbei etwas überfordert.
doch sind unsere Textverarbeitungsprogramme hierbei etwas überfordert.Die oberste Schreibweise birgt ein großes Risiko in sich, weil aus einem
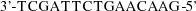 bei der laborüblichen Großzügigkeit schnell mal ein
bei der laborüblichen Großzügigkeit schnell mal ein
 wird, das mit der Originalsequenz
wird, das mit der Originalsequenz
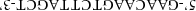 bzw.
bzw.
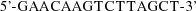 nicht mehr viel zu tun hat.
nicht mehr viel zu tun hat. -
DNA besteht fast immer aus zwei komplementären Strängen, RNA ist fast immer einzelsträngig. Das bedeutet aber nicht, dass Paarungen bei RNA nicht vorkämen, ganz im Gegenteil, nur finden sie dort innerhalb ein und desselben Stranges statt und sind so von großer Bedeutung für die dreidimensionale Struktur einer RNA, während man sich doppelsträngige DNA als ein lineares Molekül vorstellen muss.
-
Haben Sie den Fehler bemerkt? Doppelsträngige DNA besteht natürlich nicht aus einem Molekül, sondern aus zweien, Strang und Gegenstrang. Die beiden können jederzeit voneinander getrennt werden, wenn man nur genügend Energie zuführt, man spricht dann von Denaturieren. Zehn Sekunden bei 95 °C reichen bereits aus für eine vollständige Trennung der beiden Stränge. Wenn sie sich wieder treffen, können sie sich aber auch wieder paaren, man bezeichnet das dann als Hybridisieren oder neudeutsch als Annealen. Das Ganze ist (fast) beliebig reversibel.
DNA wird in der populären Presse auch gerne als Molekül des Lebens bezeichnet. Wieso so ein großes Wort für ein sehr langes, aber auch sehr langweiliges Molekül?
Die Natur – wen immer man sich darunter vorstellen mag – hat aus einer seltsamen Laune heraus die Eigenschaften von Nucleinsäuren optimal genutzt, um daraus eine verwirrende Vielfalt von Leben zu schaffen, und das geht so: Weil sich die Basen paaren, kann man zu einer einzelsträngigen DNA einen komplementären Strang synthetisieren, zu dem man ebenfalls wieder einen komplementären Strang synthetisieren kann, der mit dem ersten Strang identisch ist. Auf diese Weise kann DNA beliebig vermehrt werden, unter Wahrung der Reihenfolge ihrer Basen. Der Vorgang wird von speziellen Enzymen, sogenannten DNA-Polymerasen , ausgeführt und als Replikation bezeichnet.
Weil sich RNA und DNA chemisch so ähnlich sind, kann man von einem DNA-Strang auch ein (komplementäres) RNA-Molekül synthetisieren. Erledigt wird das von RNA-Polymerasen und der Vorgang heißt Transkription.
Dank eines extrem komplizierten Apparates aus sehr vielen Molekülen, den man als Ribosom bezeichnet, kann man die Sequenzinformation, die in einer RNA steckt, zur Synthese eines Proteins nutzen; dieser Vorgang heißt Translation. Proteine sind lange Polymere, deren Synthese ein bisschen wie die der Nucleinsäuren verläuft, d. h. an eine Aminosäure wird eine zweite gehängt, an die eine dritte usw., bis man am Ende ein Polypeptid erhält, landläufig auch als Protein bezeichnet. Nun gibt es 20 verschiedene Aminosäuren, aber nur vier Basen, wie kann also in einer RNA die Information für ein Protein stecken? Der Trick besteht darin, dass je drei Basen die Information für eine Aminosäure tragen. Man hat es mit einem Code zu tun, der in fast allen lebenden Zellen auf dieser Erde gleich interpretiert wird: AAA bedeutet Lysin, CAA Glutamin usw. Insgesamt gibt es 64 Triplettcodes (= 43), die für 20 verschiedene Aminosäuren und drei Stopcodons codieren – man sieht daran, die Sache ist etwas redundant. Tatsächlich bedeuten sowohl AAA als auch AAG Lysin, und wenn man alle 64 Triplettcodes durchsieht, stellt man fest, dass häufig die ersten zwei Basen über die Aminosäure entscheiden, die eingebaut werden soll, während die dritte ohne Bedeutung ist.Footnote 2 Das ist für den Molekularbiologen sehr nützlich, weil er auf diese Weise DNA-Sequenzen mutieren kann, ohne deren codierenden Eigenschaften zu verändern.
Doch genug der Proteine und zurück zu den schönen Dingen des Lebens, der DNA. Nicht jede DNA-Sequenz codiert auch für ein Protein, tatsächlich sind in unseren Chromosomen über 90 % der vorhandenen Sequenzen ohne erkennbare Bedeutung. Man streitet sich derzeit noch darüber, ob man sie als Abfall oder als Elemente mit unbekannter Funktion ansehen soll, doch wollen wir uns lieber mit dem kläglichen Rest auseinandersetzen.
Dieser Rest sind die Gene , ein imaginärer Abschnitt auf dem langen DNA-Molekül, der aus einem regulatorischen und einem transkribierten Bereich besteht. Deren Funktion erklärt sich aus dem Namen: Der regulatorische Bereich kontrolliert, in welchen Lebenslagen der andere Bereich transkribiert wird und wann nicht. Der transkribierte Bereich wiederum kann in zwei Arten von Unterbereichen eingeteilt werden, Exons und Introns . Diese Einteilung hat mit einer Eigenheit der Transkription von höheren Organismen (höher heißt in diesem Fall alles, was kein Bakterium ist) zu tun, dem Spleißen . Die transkribierte RNA wird nämlich gleich nach ihrer Synthese von einem komplexen Apparat namens Spleißosom bearbeitet; bei diesem Vorgang werden Teile der RNA-Sequenz herausgeschnitten und der Rest wieder miteinander verknüpft, eine komplizierte Geschichte, an deren Ende eine fertig prozessierte RNA steht, die um einiges kürzer ist als zuvor. Die Sequenzen, die während des Spleißens herausgeschnitten werden, nennt man auf DNA-Ebene Intron, diejenigen, die erhalten bleiben, Exon. Was von der RNA übrig geblieben ist, kann man nochmals einteilen in einen Bereich, der als Informationsträger für die Synthese eines Proteins dient, auch als codierender Bereich oder offenes Leseraster (open reading frame, ORF ) bezeichnet, und jeweils einen Bereich am 5'- und am 3'-Ende der RNA, die als nicht-codierende Bereiche (untranslated region oder UTR ) bezeichnet werden und deren Funktion erst in den nächsten Jahren verstanden werden wird. Außerdem besitzt die RNA an ihrem 5'-Ende ein methyliertes G-Nucleotid (5'-Cap) und am 3'-Ende einen Poly-A + -Schwanz , das ist eine Sequenz von 100–250 Adenosinen, Footnote 3 die nicht auf der DNA codiert sind und ein Signal darstellt, das so viel bedeutet wie „Hallo, ich bin eine messenger RNA Footnote 4 ich codiere für ein Protein“ (Abb. 1.2).
Das Eukaryotengen und seine Bestimmung. Das Gen besteht aus einem regulatorischen Bereich mit Enhancern (Enh) und Promotor (Prom) und einem transkribierten Bereich, der in RNA übersetzt wird. Die heterogene Kern-RNA (hnRNA) ist eine 1:1-Kopie der DNA mit einem Poly-A+-Schwanz (AAA) und enthält noch Exons (E) und Introns (I), doch noch im Zellkern werden die Introns durch den Spleißosom-Komplex herausgeschnitten, auf diese Weise entsteht die Boten-RNA (mRNA). Nur das offene Leseraster (ORF) der mRNA wird in Protein translatiert, während die nichttranslatierten Bereiche (UTRs) am 5'- und am 3'-Ende teils für die Stabilität der RNA, teils für ihre Lokalisation, teils für gar nichts verantwortlich sind
All diese Bereiche sind, wohlgemerkt, imaginär, man sieht einer DNA-Sequenz nicht an, ob sie aus dem Exon eines Gens stammt oder eine der vielen Abfallsequenzen ist, zumindest im Augenblick noch nicht. Vielleicht ändert sich das eines Tages, zumindest arbeiten gegenwärtig viele schlaue Leute daran, genau diesen Zustand zu ändern. Mal sehen, ob sie erfolgreich sein werden, sie würden uns allen viel Arbeit ersparen und den Molekularbiologen überflüssig machen.
So viel in allerkürzester Kürze zu den Grundlagen von Molli-World. Darüber hinaus hat der Experimentator auch noch mit einer Handvoll Proteinen zu tun, mit denen er seine Nucleinsäuren bearbeitet – Polymerasen, Restriktionsenzyme, Kinasen, Phosphatasen usw. – und von denen er nicht so recht versteht, wie sie funktionieren, was im Grunde auch nicht notwendig ist, solange sie funktionieren. Sollte das nicht der Fall sein, bestellt er meistens einfach neue, weshalb es sich erübrigt, hier genauer auf sie einzugehen.
1.2 Was brauche ich zum Arbeiten?
Einen Arbeitsplatz in einem Labor mit einer Genehmigung für gentechnische Versuche, drei Pipetten, mit denen man Volumina zwischen 0,5 und 1000 µl pipettieren kann, der Rest ist Luxus. Hat man zumindest den Eindruck.
Man sollte seine Erwartungen nicht zu hoch schrauben, die meisten Arbeitsgruppen haben hohe Ansprüche und eine geringe Ausstattung. Schon einen Schreibplatz bekommt man häufig nicht, oder man muss ihn sich mit jemandem teilen. Der Rest ist entsprechend, so dass man nehmen sollte, was man bekommen kann, um dann zu versuchen, das Beste daraus zu machen.
Man braucht jede Menge Flaschen für seine Puffer, Messzylinder und Glaspipetten, Zentrifugen und Chemikalien, Kühl-, Gefrier- und Tiefgefrierschränke, PCR-Maschinen und Waagen – sprich: Wer noch kein Profi auf dem Gebiet ist, sollte besser die Finger davon lassen, ein Labor etablieren zu wollen, wenn er nicht ein Jahr seines Lebens verlieren möchte.
Wie sollte der eigene Arbeitsplatz aussehen? Naja, im Prinzip sieht's natürlich jeder gerne, wenn der Arbeitsplatz ordentlich ist, denn Ordnung ist das halbe Leben. Meint Lieschen Müller. Eine andere Überlegung allerdings besagt, dass ein chaotischer Arbeitsplatz, an dem sich kein Mitarbeiter befindet, wenigstens so aussieht, als würde daran gearbeitet, während eine glänzende, gähnend leere Fläche kaum einen Chef davon überzeugen dürfte, dass man einen großen Arbeitseifer an den Tag legt.
Grundsätzlich kann man Labormenschen in Chaoten und Pingelmänner einteilen. Finden Sie selbst heraus, wozu Sie gehören. Beides ist genetisch festgelegt und nur in Maßen zu beeinflussen, verschwenden Sie daher nicht Ihre Zeit damit, Ihren Kollegen ändern zu wollen, es klappt eh nicht. Lernen Sie, mit der Situation zu leben – was nicht heißt, dass man sich alles gefallen lassen müsste. Manche Kollegen neigen beispielsweise dazu, sich ausgiebig der Lösungen anderer Leute zu bedienen, weil sie zu faul sind, selber welche anzusetzen, so dass man regelmäßig in der größten Versuchshektik vor einer leeren Pufferflasche steht. Ein beliebtes Gegenmittel ist das Umetikettieren von Flaschen nach dem Motto „Wo Tris draufsteht, ist garantiert kein Tris drin“, nicht legal, aber wirkungsvoll, solange der Kollege das System nicht kennt, nach dem umetikettiert wurde. Diese Methode eignet sich allerdings bestenfalls als Notbremse für extrem uneinsichtige Zeitgenossen, weil es auch das Verhältnis zu den anderen Kollegen nicht verbessert, außerdem hat man sein eigenes System spätestens nach dem Urlaub vergessen …
Wie sollte man arbeiten? Viele pflegen mit großer Überzeugung das Prinzip des kreativen Chaos. Das geht solange gut, bis man in seinem Kühlschrankfach fünf Ständer mit Tubes vorfindet, die man in den letzten drei Wochen akkumuliert hat und die alle mit 1 bis 24 durchnummeriert sind. Man sollte sich daher von Anfang an ein gewisses System bei seiner Arbeit angewöhnen, damit man in den „heißen“ Phasen zumindest mit scharfem Überlegen noch nachvollziehen kann, was man gemacht hat.
Auch sollte man sich mit einer einigermaßen sauberen Arbeitsweise anfreunden. Nucleinsäuren sind recht stabile Moleküle, die nur eines fürchten: Nucleasen. Leider sind Nucleasen ein bisschen überall, angefangen bei unseren Fingern. Wer schlampig arbeitet, findet sich bald mit einer Plasmid-DNA-Präparation wieder, die aus vielen kleinen DNA-Stückelchen besteht, mit denen er nichts mehr anfangen kann. Daher autoklaviert man üblicherweise alles, womit die DNA in Berührung kommen könnte: Pipettenspitzen, Reaktionsgefäße, Glasflaschen, Lösungen … Vieles davon kann man sich sparen, wenn man erst einmal gelernt hat, sauber (d. h. nucleasefrei) zu arbeiten. Beispielsweise sind die Plastikreaktionsgefäße bei Lieferung praktisch immer nucleasefrei, selbst wenn's nicht draufsteht, und müssen nicht unbedingt autoklaviert werden, wenn es einem gelingt, sie aus der Tüte zu kippen, ohne sie anzufassen. Doch damit sollte man erst anfangen, wenn man sich einigermaßen sicher fühlt bei dem, was man macht. Die Nagelprobe ist der Umgang mit Bakterienmedium: Wem es gelingt, eine Woche lang ein und dieselbe Flasche mit LB-Medium zu benutzen, ohne dass sich darin Bakterien oder Hefen breit machen, der hat's gelernt. Bedenken Sie aber in jedem Fall, dass auch die ausgeprägteste Autoklavierwut verlorene Liebesmüh ist, wenn Sie anschließend beispielsweise mit Ihren schmutzigen Fingerlein im Glas mit den sterilen Eppendorf-Tubes herumwühlen!
Eine andere Sache, die man sich angewöhnen sollte, ist das Führen eines Laborbuches . Erstens braucht man es, um den Überblick zu wahren, zweitens ist es Vorschrift. Niemals länger als eine Woche mit dem Nachtragen warten, weil man bis dahin schon die Hälfte der Details vergessen hat. Am besten ist es, diese Arbeit jeweils am Ende eines langen erfolgreichen Arbeitstages, vielleicht bei einem Tässchen Kaffee, zu erledigen und sich gleichzeitig in Ruhe zu überlegen, was man am nächsten Tag tun will oder muss – ich bin mir allerdings dessen bewusst, dass dieser Vorschlag so realistisch ist wie der Wunsch, ab morgen mögen alle Menschen gut sein.
1.3 Sicherheit im Labor
Im molekularbiologischen Labor hat man es mit einer ganzen Reihe von Sicherheitsverordnungen zu tun. Viele davon haben mit Biologie nicht viel zu tun. So gelten beispielsweise dieselben Richtlinien wie in chemischen Labors, die sich vor allem mit den Fragen sicheren Arbeitens und des Unfallschutzes beschäftigen. Sie verbieten beispielsweise Essen, Trinken und Rauchen im Labor und verpflichten zum Tragen eines Labormantels, von festen, geschlossenen, trittsicheren Schuhen (d. h. keine Sandalen, keine Stöckelschuhe usw.) und einer Schutzbrille (jawohl, den ganzen Tag lang). Im Gegensatz zu früher reicht eine normale Brille übrigens nicht mehr, angeblich soll stattdessen der Arbeitgeber verpflichtet sein, eine Sicherheitsbrille mit Korrekturgläsern zur Verfügung zu stellen. Man darf sich zwar nicht wundern, wenn solche Anweisungen von Leuten, die den lieben langen Tag Kleinstmengen von Salz- und Proteinlösungen von einem Plastikgefäß ins andere pipettieren, zumeist ignoriert werden, trotzdem ist natürlich jeder angehalten, sich danach zu richten.
Beim Umgang mit gefährlichen Substanzen sind außerdem Handschuhe zu tragen – einerseits zum Schutz der eigenen Fingerchen, aber auch zum Schutz der Mitmenschen. Es reicht daher nicht, Handschuhe anzuziehen, wenn man beispielsweise mit Ethidiumbromid arbeitet, man muss sie auch wieder ausziehen, wenn man nicht mehr damit arbeitet, um nicht an Türklinken, Telefonhörern und Wasserhähnen hauchdünne Giftfilme zu hinterlassen.
A propos Handschuhe: Man kann nicht ohne sie sein, aber mit ihnen ist es auch nicht leicht. Am untauglichsten sind im Labor Vinylhandschuhe – sie sind schwierig anzuziehen, passen schlecht und bekommen leicht Löcher. Wesentlich besser sind Latexhandschuhe, die es in verschiedenen Größen und Stärken gibt. Sie liegen wie eine zweite Haut an, sind aber leider allergen, vor allem die gepuderte Variante, von der man entschieden abraten muss, weil sich im Laufe der Monate und Jahre bei den meisten Leuten Hautprobleme einstellen. Hat man erst einmal eine satte Latexallergie, kann sogar das Puder von den Handschuhen des Nachbarn zum Problem werden. Die neueste Entwicklung sind Nitrilhandschuhe, die nicht ganz so elastisch wie Latex und auch ein wenig teurer sind, aber dafür das geringste Allergierisiko beinhalten.
Die zweite Gruppe von Sicherheitsverordnungen betrifft den Umgang mit radioaktiven Substanzen. Deren Handhabung ist grundsätzlich verboten, es sei denn, das Institut besitzt eine Genehmigung für den Umgang. Das Vorhandensein einer Genehmigung bedeutet aber nicht, dass man einfach loslegen könnte. Der Weg führt zwangsläufig über den Strahlenschutzbeauftragten, der Ihnen dann im Detail erklären wird, mit welchem Radionuclid und welchen Mengen gearbeitet werden darf, in welchen Räumen und unter Einhaltung welcher Sicherheitsmaßnahmen.
Die biologische Sicherheit schließlich wird über die Gentechnik-Sicherheitsverordnung (Gen-TSV) Footnote 5 geregelt. Die legt fest, dass die Verantwortung beim Projektleiter liegt, das gilt einerseits für den administrativen Teil und andererseits auch für die Einweisung und Beaufsichtigung der Beschäftigten. Projektleiter kann nur werden, wer ein abgeschlossenes (naturwissenschaftliches oder medizinisches) Hochschulstudium und mindestens drei Jahre Tätigkeit auf dem Gebiet der Gentechnik vorzuweisen hat, außerdem muss er eine Fortbildungsveranstaltung zu den Themen gentechnisches Arbeiten, Sicherheitsmaßnahmen und Rechtsvorschriften besucht haben.
Trotzdem ist die Gentechnik-Sicherheitsverordnung natürlich auch für den Jungforscher interessant. Sie regelt die Einteilung der (gentechnisch veränderten) Organismen in Risikogruppen , wobei, grob gesagt, zur Risikogruppe 1 all jene Mikroorganismen und Zellkulturarbeiten gehören, die kein Risiko darstellen, während Gruppe 2 einem geringen, Gruppe 3 einem mäßigen und Gruppe 4 einem hohen Risiko entspricht. Zum Glück obliegt die Zuordnung nicht dem eigenen Gutdünken, sondern erfolgt durch die Zentrale Kommission für die Biologische Sicherheit (ZKBS) . So gehört E. coli K12, der Urahn der meisten im Labor verwendeten Bakterienstämme, zur Risikogruppe 1, ebenso wie beispielsweise die Labormaus, Drosophila melanogaster oder die Zelllinien CHO, PC12 oder HeLa. In den höheren Risikogruppen findet man hauptsächlich Viren wie Adenovirus (Gruppe 2), Hepatitis B Virus (Gruppe 2), Hepatitis C Virus (Gruppe 3) oder HIV (Gruppe 3) und Zelllinien, die derartige Viren abgeben können. Footnote 6
Den Risikogruppen sind entsprechende Sicherheitsstufen zugeordnet, die vorschreiben, in welchen Räumlichkeiten die jeweiligen Experimente durchgeführt werden dürfen. So muss ein Labor der Sicherheitsstufe 1 vier Wände, eine Decke, Fenster und wenigstens eine Tür besitzen, wobei Letztere geschlossen zu halten ist. Es darf keine Abstellkammer sein („Die Arbeiten sollen in abgegrenzten und in ausreichend großen Räumen bzw. Bereichen durchgeführt werden“) und ist als Gentechnik-Arbeitsbereich zu kennzeichnen. Die Liste der absonderlich wirkenden Vorschriften ist noch erheblich länger, so muss das Labor ein Waschbecken besitzen, an dem man sich feierabends die Hände wäscht, nachdem man zuvor den Raum aufgeräumt und das Ungeziefer bekämpft hat. Andere Anordnungen sind dagegen schon aus anderen Verordnungen bekannt. So muss man einen Laborkittel tragen und darf nicht mit dem Mund pipettieren. Interessanterweise sind Essen, Trinken, Rauchen und Schminken zwar verboten, das Aufbewahren von Nahrungs- und Genussmitteln sowie Kosmetika jedoch gestattet, sofern sie „mit gentechnisch veränderten Organismen nicht in Berührung kommen“.
Ernsthafter wird es in Labors der Sicherheitsstufe 2. Schon optisch, weil diese mit einem Warnzeichen Biogefährdung zu kennzeichnen sind, beim Waschbecken ein Desinfektionsmittel stehen soll und neben den Türen nun auch die Fenster geschlossen sein müssen. Es dürfen keine Aerosole freigesetzt werden, die Arbeitsflächen sind zu desinfizieren und alles, was mit gentechnisch veränderten Organismen (GVOs) in Kontakt kommt, muss autoklaviert bzw. desinfiziert werden. Außerdem soll der Betreiber Schutzkleidung bereitstellen. Betreten darf solche Labors nur, wer dazu ausdrücklich befugt ist.
Bei Labors der Sicherheitsstufe 3 braucht man die Fenster nicht geschlossen zu halten, weil die Verordnung vorsieht, dass man sie gar nicht öffnen kann (bzw. können darf). Man kann ein solches Labor nur über eine Schleuse betreten, in der man dann eine geeignete Schutzkleidung anlegen muss, außerdem muss dort ein Autoklav stehen, das Labor muss unter Unterdruck stehen und man darf dort nicht alleine arbeiten.
Ein Labor der Stufe 4 schließlich gleicht im Großen und Ganzen einem Bunker, in dem sich als Astronauten verkleidete Lebensmüde tummeln. Die Liste der vorgeschriebenen Sicherheitsmaßnahmen ist mit zwei Seiten immerhin doppelt so lang wie bei den anderen Sicherheitsstufen und nimmt einem die Lust am Arbeiten. Weltweit ist die Zahl der S4-Laboratorien mit ca. dreißig noch ziemlich überschaubar, allerdings ist die Tendenz eindeutig steigend. Bestes Beispiel dafür ist Deutschland: Gab es hier bis vor wenigen Jahren noch gar kein S4-Labor, hat 2007 das erste in Marburg (Philipps-Universität) seine Pforten geöffnet,Footnote 7 ein zweites ist 2008 in Hamburg (Bernhard-Nocht-Institut) in Betrieb gegangen und auf der Insel Riems (Friedrich-Loeffler-Institut) sowie in Berlin (Robert-Koch-Institut) ist jeweils eines im Bau.
Auch die Entsorgung biologischer Abfälle und Abwasser wird durch die Gentechnik-Sicherheitsverordnung geregelt. Sie kann bei Anlagen der Sicherheitsstufe 1 ohne Vorbehandlung erfolgen, sofern keine Vermehrung bzw. Infektionsgefahr zu erwarten ist. Erst ab Sicherheitsstufe 2 müssen solche Hinterlassenschaften zwingend inaktiviert bzw. autoklaviert werden. Ungeachtet dieser großzügigen Regelung sollte man es sich in jedem Fall zur Gewohnheit machen, Bakterienkulturen, Schalen aus der Zellkultur, DNA- und bakterienverschmutzte Pipettenspitzen und ähnliche Nebenprodukte produktiven Schaffens vor dem Entsorgen zu autoklavieren, um sich nicht irgendwann von irgendjemandem liederliches Arbeiten vorwerfen lassen zu müssen. Dies entspricht auch den Regeln der Good Laboratory Practice (GLP ), wie sie durch die Organization for economic co-operation and development (OECD) festgelegt worden sind. Dieses Werk regelt auch ganz andere Bereiche des Laborlebens (beispielsweise wie man seine Versuche dokumentiert) und wird auch in Deutschland als Richtlinie für das ordentliche Arbeiten im Labor angesehen. Wer sich für GLP interessiert, kann sich übers Internet informieren (beispielsweise unter http://www.oecd.org/ – suchen Sie dort nach GLP).
Auch zum Thema Gentechnik und Sicherheit kann man sich übers Internet schlauer machen. Das Robert-Koch-Institut, sozusagen Deutschlands höchste Instanz auf diesem Gebiet, veröffentlicht im Internet nützliche Informationen wie beispielsweise die Liste der durch die ZKBS bewerteten Organismen und Vektoren oder allgemeine Stellungnahmen der ZKBS zu gentechnikrelevanten Themen und vieles andere mehr (siehe http://www.rki.de unter dem Stichwort Gentechnik). Oder man wende sich an den örtlichen Beauftragten für die Biologische Sicherheit.
Notes
- 1.
Die Faust-Zitate sind der dtv-Gesamtausgabe von 1966 (3. Aufl.) entnommen, die auf der „Gedenkausgabe der Werke, Briefe und Gespräche“ des Artemis-Verlags beruht.
- 2.
Sehr interessant in diesem Zusammenhang ist eine Publikation von Roland Pohlmeyer (2008). Viele kennen die Darstellung des genetischen Codes als Code-Sonne, die auf Bresch und Hausmann (1972) zurückgeht und die Redundanz des Codes sehr anschaulich macht. Obwohl für die Praxis sehr nützlich, hinterlässt die Code-Sonne allerdings ein Fragezeichen, weil sie 25 Gruppen aufweist, obwohl nur 21 benötigt würden (für 20 Aminosäuren und die Stopcodons). Pohlmeyer zeigt, dass für die Logik (und die Evolution) des Codes eigentlich die zweite Base die entscheidende ist. Sortiert man die Codes zunächst nach der zweiten und erst dann nach der ersten und schließlich dritten Base, erhält man eine perfekte Code-Sonne mit 21 Gruppen, bei der nicht nur erstmals alle Codes für Leucin, Arginin und Serin nebeneinanderliegen, sondern auch Aspartat, Asparagin, Glutamat und Glutamin benachbart sind. Und dass einige Abweichungen, die man bei den Nicht-Standard-Codes findet, in dieser Anordnung plötzlich nicht mehr völlig willkürlich erscheinen, ist zumindest ein interessante Feststellung. Mir scheint, dass die modifizierte Code-Sonne der Evolutionsforschung einige interessante Denkanstöße liefern kann.
- 3.
Die Literaturangaben hierzu schwanken recht stark.
- 4.
Messenger RNA (mRNA ) oder Boten-RNA, wie man im Deutschen mitunter sagt, ist nur eine von drei Hauptklassen an RNA; mengenmäßig von geringer Bedeutung (sie macht nur ca. 2 % der gesamten RNA einer Zelle aus), spielt sie dennoch eine wichtige Rolle, weil die mRNAs die Sequenzinformation für die Synthese der Proteine enthalten. Daneben gibt es noch die Klasse der ribosomalen RNAs (rRNA ), die wichtiger Bestandteil der Ribosomen sind, und die Klasse der Transfer-RNA (tRNA ), kleine RNAs, an die die Aminosäuren gekoppelt werden, bevor sie dann in die Proteine eingebaut werden. Darüber hinaus enthält eine normale Zelle noch eine Reihe weiterer RNAs, von denen man allerdings selten spricht, mit Ausnahme der siRNAs (Abschn. 5.7).
- 5.
Der Langtitel lautet übrigens: Verordnung über die Sicherheitsstufen und Sicherheitsmaßnahmen bei gentechnischen Arbeiten in gentechnischen Anlagen.
- 6.
Für den Fall, dass Sie sich fragen, welche Art von Organismen wohl in die Risikogruppe 4 fallen mögen: zum Beispiel Ebola-, Pocken-, Lassa-, SARS- oder Marburg-Virus.
- 7.
…und hoffentlich seine Schleusen geschlossen.
Literatur
Ashbrook PC, Renfrew MM (1990): Safe Laboratories: Principles and Practices for Design and Remodelling. Lewis Publishers
Bresch C, Hausmann R (1972) Klassische und molekulare Genetik, 3. Auflage. Springer Verlag
Block S (1991): Disinfection, sterilization and preservation. Lea & Febinger
Flemming DO et al. (Hrsg.) (1995): Laboratory safety. Principles and practices. American Society for Microbiology
Pohlmeyer R (2008) The genetic code revisited. J. Theor. Biol. 253, 623–624
Rayburn SR (1990): The Foundations of Laboratory Safety. Springer-Verlag
Author information
Authors and Affiliations
Rights and permissions
Copyright information
© 2013 Springer-Verlag Berlin Heidelberg
About this chapter
Cite this chapter
Mülhardt, C. (2013). Was bitte ist denn »Molekularbiologie«?. In: Der Experimentator Molekularbiologie/Genomics. Der Experimentator. Springer Spektrum, Berlin, Heidelberg. https://doi.org/10.1007/978-3-642-34636-1_1
Download citation
DOI: https://doi.org/10.1007/978-3-642-34636-1_1
Publisher Name: Springer Spektrum, Berlin, Heidelberg
Print ISBN: 978-3-642-34635-4
Online ISBN: 978-3-642-34636-1
eBook Packages: Life Science and Basic Disciplines (German Language)




 Genau genommen ist diese Schreibweise falsch, weil die beiden DNA-Stränge gegenläufig zueinander angeordnet sind. Korrekterweise
müsste man die obige Sequenz folglich so darstellen
Genau genommen ist diese Schreibweise falsch, weil die beiden DNA-Stränge gegenläufig zueinander angeordnet sind. Korrekterweise
müsste man die obige Sequenz folglich so darstellen
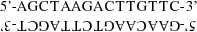 doch sind unsere Textverarbeitungsprogramme hierbei etwas überfordert.
doch sind unsere Textverarbeitungsprogramme hierbei etwas überfordert.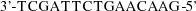 bei der laborüblichen Großzügigkeit schnell mal ein
bei der laborüblichen Großzügigkeit schnell mal ein
 wird, das mit der Originalsequenz
wird, das mit der Originalsequenz
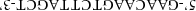 bzw.
bzw.
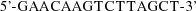 nicht mehr viel zu tun hat.
nicht mehr viel zu tun hat.
